大規模データの高速な共有と解析を実現する
広域分散ファイルシステム「Gfarm」
 筑波大学計算科学研究センターは「計算科学」と「計算機科学」という2つの分野の研究者が車の両輪となって互いに連携しながら、研究活動を推進している施設です。今回、紹介する建部修見(たてべ・おさむ)さんは、そのうちの計算機科学の研究者で、超高速計算システム、グリッドコンピューティング、並列分散システムソフトウェアの研究開発を専門としています。2002年に建部さんが中心となって開発した「Gfarm」は、大量のデータを、国内外の研究者同士が共有したり、データ解析したりするための「広域分散ファイルシステム」です。以下ではGfarmの特徴と開発の歴史を紹介していきましょう。
筑波大学計算科学研究センターは「計算科学」と「計算機科学」という2つの分野の研究者が車の両輪となって互いに連携しながら、研究活動を推進している施設です。今回、紹介する建部修見(たてべ・おさむ)さんは、そのうちの計算機科学の研究者で、超高速計算システム、グリッドコンピューティング、並列分散システムソフトウェアの研究開発を専門としています。2002年に建部さんが中心となって開発した「Gfarm」は、大量のデータを、国内外の研究者同士が共有したり、データ解析したりするための「広域分散ファイルシステム」です。以下ではGfarmの特徴と開発の歴史を紹介していきましょう。
データの格納場所を意識することなくストレージに高速にアクセス可能
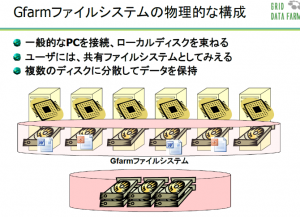
近年、大規模実験施設で観測されたデータやスパコンによる大規模シミュレーションによって得られたデータの容量が増え続けています。それに伴い、大規模データを、国内外を問わず、広域にわたり多くの研究者同士で共有したり、解析したいといったニーズが高まっています。そのニーズに応えるべく建部さんが中心となり開発したのがGfarmです。図1はGfarmの構成をわかりやすく図式化したものです。ファイルシステム全体は広域に設置された複数のストレージで構成されています。これらのストレージを1つに束ねて、あたかも1つのファイルシステムのように見せているのが、Gfarmファイルシステムです。データは実際には複数のストレージに分散して格納されていますが、ユーザー側からは1つのデータのかたまりとして見えています。そして、どのコンピュータからでも、データの格納場所をまったく意識することなく、ストレージに高速にアクセスすることができるのです。これがGfarmの最大の特徴です。
Gfarmの特徴はほかにもたくさんあります。1つめは、システムの運用を停止することなく、ストレージをいくらでも増設できることです。2つめは、データの信頼性が高いことです。通常のファイルシステムの場合、ストレージにデータを大量に格納していると、「サイレントデータ損傷」と呼ばれる障害が発生することがあります。これは、エラーメッセージが出ることなく、データがいつの間にか損傷してしまう障害です。それに対し、Gfarmでは、データの読み書きの都度、データをチェックするため、サイレントデータ損傷を検出し修復します。
さらに、複数の拠点間でのデータの複製機能、ユーザーがより近い拠点のデータに自動的にアクセスする機能、データ解析の際にローカルディスクを用いる機能など、データへのアクセスと解析を高速化する機能が実装されていることも大きな特徴です。これらについては、あとで実際の応用に即して詳しく説明します。
世界で初めて広域の高速データ転送を達成
世界規模のデータセンターの実現に目途
そもそも建部さんがGfarmの研究開発に着手したのは2000年のこと。きっかけは、その10年後に開始予定だったCERNの大型ハドロン衝突型加速器LHCを使った素粒子に関する大規模な実験プロジェクトでした。
この実験プロジェクトでは、1年間に約1ペタバイト(ペタは1015)という非常に大規模な実験データを生成し、それを世界中のスパコンを使って解析するという壮大な計画を立てていました。この計画を実現するには、データを広域にわたって共有するための新たなシステムを開発する必要がありました。そこで、当時、電子技術総合研究所(電総研)に在籍していた建部さんは、LHCの大規模実験に貢献すべく、高エネルギー加速器研究機構(KEK)と共同で、日本独自のシステムの研究開発に着手したのです。
そして、2002年11月に米国で開催された国際会議SC2002において、開発したファイルシステムを用いて実証実験を行いました。まず、日本とアメリカの7拠点に設置された190台のPCのハードディスクを高速ネットワークで接続。18テラバイト(テラは1012)の大規模データをこれらのハードディスクに分散して格納。さらに分散して格納したデータ同士を、システムを使ってすべて協調させ、解析できるようにしました。大規模データの転送やデータへのアクセスを並列処理することで、高速化を実現しました。
この実験では、1万キロメートルも離れた日米間において、世界で初めて741メガbpsというデータ転送速度を達成。この成功により、世界規模の大規模データセンターの実現や、国際的な共同実験による大規模データ解析が実現可能であることが、初めて示されたのです。
さらに建部さんたちは2003年11月開催のSC2003で、並列分散による別の大規模データ解析実験を成功させ、「分散インフラストラクチャ賞」を受賞しました。そして同年、建部さんは、このファイルシステムをGfarmとして広く公開したのです。
素粒子宇宙研究のデータグリッド「JLDG」にも搭載
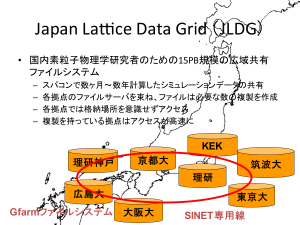
さらに、2005年から建部さんは筑波大学計算科学研究センター素粒子宇宙研究部門の吉江友照さんたちと共同で、Gfarmを搭載したデータグリッド「JLDG(Japan Lattice Data Grid)」(図2)の開発を進めました。
素粒子宇宙分野では、格子QCDシミュレーションによるハドロン物理や素粒子標準模型の研究などが進められています。しかし、格子QCDシミュレーションの場合、計算が非常に大規模です。そこで、2002年に複数の研究機関のスパコンを使って基礎データを生成し、これをネットワークを介して共有しようというプロジェクトが発足しました。その結果、筑波大学を含む全国6つの研究拠点のストレージ間のミラーリング(同じデータを書き込むこと)によるデータ共有が始まりました。このシステムでは、ひとかたまりとして利用するデータが複数のハードディスクに分散して格納されており、ユーザー側でそれぞれのデータの所在を意識する必要がありました。さらに、「データの所在を把握し切れない」、「サポート体制が整っていないため、障害が発生した場合、迅速に対応できない」といった課題を抱えていました。そこで、これらの課題を解決するため、新たに開発されたのがJLDGだったのです。JLDGは2008年5月に正式運用が開始されました。
JLDGのファイルシステムにGfarmを採用したことで、ユーザーはデータの所在を意識することなくデータ共有ができるようになったほか、組織をまたぐ一元的なユーザー管理が可能になり、従来のミラーリングによるデータ共有が抱えていた課題をすべて解決することができました。
現在、JLDGでは、筑波大学、KEK、理研本部(和光)、理研R-CCS(神戸)、東京大学、京都大学、大阪大学、広島大学の8拠点においてスパコンで数カ月~数年間にわたり計算した格子QCDのシミュレーションデータを共有しており、各拠点では、ユーザーは自分が所属する組織のサーバにログインするだけで、データの所在を意識することなく、研究に必要なデータに自由にアクセスできるようになっています。また、Gfarmのデータの複製機能により、複製を保有している拠点ではさらなる高速アクセスが可能となっています。
一方で、国際的なデータグリッドとしては、「ILDG(International Lattice Data Grid)」が公開されています。JLDGも欧米やオーストラリアのデータグリッドとともにILDGに参加しており、各国のデータグリッド同士でネットワークを形成しています。ILDGを介して国内外の素粒子物理の研究者に対し、格子QCDに関するシミュレーションデータなどを提供しています。
1000ノード超のクラスタを使い
2週間かかっていたデータ解析を30分に短縮
また、2006年に行ったKEKとの共同研究では、KEKのスパコンセンターのメインマシンに、Gfarmを最適化して導入。1,112ノードのクラスタを使い、計算ノードのローカルディスクを用いることで、全部で26テラバイトという大規模な高速ストレージシステムを構築しました。これにより、それまで1~2週間かかっていた実験データの解析を30分に短縮することに成功しました。
ここでのポイントを建部さんはこう話します。「従来のメインマシンにおいて、解析に使う計算ノードは十分足りていました。それにもかかわらず、解析に時間がかかっていたのは、データに高速にアクセスすることができなかったからです。この共同研究では、Gfarmを使い、データを計算ノードのローカルディスクに格納しました。それにより、共有ストレージシステムから読み込んでくるよりも高速にデータにアクセスできるようになりました。加えて、たとえば、1,000ノードを超えるクラスタが一斉にデータにアクセスする場合、従来であればネットワークがパンクしてしまうことが危惧されます。しかしアクセスする先がローカルディスクなので、ネットワークがパンクする心配はありませんでした。その結果、データ解析にかかる時間を大幅に短縮できたのです」
HPCIの共用ストレージにも採用
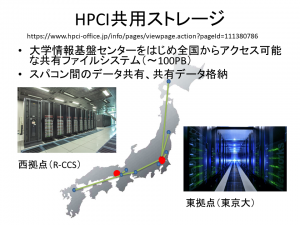
図3:HPCIは、スーパーコンピュータ「富岳」と国内の9大学(北海道大学、東北大学、筑波大学、東京大学、東京工業大学、名古屋大学、京都大学、大阪大学、九州大学)と国立研究所のスパコンからなる。その共用ストレージは東京大学情報基盤センター(東拠点)と理化学研究所計算科学研究センター(R-CCS)(西拠点)に設置され、Gfarmが採用されている。写真提供:東京大学情報基盤センター(撮影:三浦健司)、理化学研究所
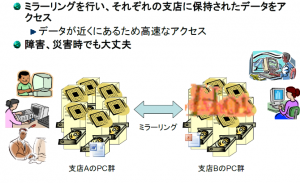
東大とR-CCSのストレージには実はそれぞれ同じデータが複製(ミラーリング)され、格納されています。複製を格納していることのメリットは2つです。1つ目は、障害に対する備え、2つ目は、アクセス性能の向上です(図4)。「万が一、片方のストレージで障害が発生しても、もう一方のストレージに自動的にアクセスできるようになっているため、ユーザーは障害を意識することなくデータを参照することができます。また、たとえば、東京のユーザーがアクセスすると、自動的に東大のストレージにアクセスするといった具合に、より距離が近いストレージにアクセスするようになっています。それにより、高速なデータアクセスを実現しているのです。この機能は特に広域でデータを共有する際に威力を発揮します。多くの方に快適に利用していただくため、高いアクセス性能とデータの信頼性を最も重要視しています」と建部さん。
さらに近年は、理論、実験、計算科学に次ぐ第4の科学として、データ科学の重要性が高まっています。データ科学では人工知能(AI)を使い、いかに高速にビッグデータを解析するかが課題となっています。それに対し、建部さんはこう話します。「『富岳』でも、AIを使ったビッグデータ解析に力を入れています。AIによるビッグデータ解析を推進するには、CPUの性能を上げるのはもちろんですが、まずは、ビッグデータの収集と整備が不可欠です。データは大規模になればなるほど、高速転送が重要性を増していきます。その点で、Gfarmは貢献できると考えています」。また、Gfarmは、ビッグデータ解析を目的とする企業にも利用されており、ユーザー層が拡大しています。このため、建部さんはNPO法人つくばOSS技術支援センターを立ち上げ、Gfarmのユーザーサポートも行っています。
今後さらなる大規模実験や大規模シミュレーションが計画されていることに加え、AIによるビッグデータ解析の重要性が増す中、大規模データの共有や解析を高速に実現するGfarmへのニーズはますます高まっていくことでしょう。